Różowe prążki w żelu
Co Pani wie o Estonii? – usłyszałam osiem miesięcy temu w gabinecie mojego promotora. – Cóż, Estonia – piękny kraj – odrzekłam z niefrasobliwym uśmiechem. Miesiąc później, oglądając z lotu ptaka na wpół zamarznięty kawałek sosnowej puszczy, przeklinałam to wyświechtane stwierdzenie na tysiąc sposobów. Wtedy zaczęła się moja wschodnioeuropejska przygoda.
Gdy pierwszego poranka obudziłam się w akademiku, gdzie mnie zakwaterowano z dwiema rozrywkowymi Finkami, przez głowę przebiegło mi stado myśli, z których najważniejsza brzmiała: „Co ja powiem Estończykom, kiedy zapytają, co chcę tu robić?” Tak się przejęłam, że na pierwszym spotkaniu z moim estońskim opiekunem usta mi się nie zamykały. Opowiadałam, co chciałabym robić, dlaczego tak, czemu tu i po co. Mój opiekun słuchał, słuchał i... odniosłam dziwne wrażenie, że w ogóle go to nie interesuje, a jedyną jego myślą jest: „Czemu ta baba tak trajkocze?” Otóż pierwsze wrażenie myli – on po prostu był Estończykiem, ten typ tak ma.
„Europejskie standardy” to fraza często nadużywana w omawianiu działania różnych instytucji, nie tylko naukowych. Ale Instytut Biologii Komórki i Biologii Molekularnej Uniwersytetu w Tartu spełnia unijne wymogi co do joty. Po pierwszym tygodniu spędzonym w ich laboratorium byłam zachwycona. Dusza biologa śpiewała we mnie pełnym głosem.
Bakterie osadu czynnego
Mój „laboratoryjny chleb powszedni” od połowy studiów i obiekt żywego zainteresowania współczesnych nauk biologicznych to DNA – kwas dezoksyrybonukleinowy. Ta dwuniciowa, spiralnie zwinięta cząstka, zbudowana jest z podjednostek zwanych nukleotydami. Każdy nukleotyd zawiera jedną z czterech zasad azotowych – adeninę, guaninę, cytozynę lub tyminę. Dzięki genialnej w swej prostocie regule parowania się tych zasad, znając sekwencję, czyli kolejność ułożenia nukleotydów w jednej nici, możemy bez problemów odtworzyć kontinuum nukleotydów drugiej. Korzystając ze znajomości budowy kwasów nukleinowych, stworzono pełną gamę metod, pozwalających, między innymi, na identyfikację organizmów, porównywanie ich DNA, wykrywanie mutacji różnej wielkości i pochodzenia oraz analizę filogenetyczną, zajmującą się ustalaniem pokrewieństwa ewolucyjnego istot żywych.
Rozejrzyjmy się wokół. Na naszym globie występują ogromne ilości wody. Bez tej substancji nasze życie byłoby niemożliwe. Ze względu na postępujące zanieczyszczenie środowiska, zasoby czystej wody kurczą się w zastraszający sposób. Czemu więc nie zaprząc nauki do ocalenia tego, co nam jeszcze zostało? Dlatego badania, które zamierzałam przeprowadzić w Tartu, skupiały się na bakteriach osadu czynnego, czyli mieszaninie mikroorganizmów używanych do biologicznego oczyszczania wody i ścieków.
Ze względu na proekologiczną politykę większości państw europejskich metody biologiczne wykorzystywane w technologiach utylizacji odpadów stały się nie tyle modne, co konieczne. Dlatego też ważnym zagadnieniem, nie tylko z punktu widzenia technologii, jest poszukiwanie mikroorganizmów zdolnych do wydajnej eliminacji zanieczyszczeń. Pytanie: co ma do tego biologia molekularna? Otóż organizmy występujące w sztucznych układach biologicznych, jakimi są bioreaktory osadu czynnego, pozwalają śledzić ich zmienność genetyczną oraz szybkość tworzenia się nowych „rozwiązań metabolicznych” w postaci uruchamiania wydajnych szlaków rozkładu związków szkodliwych. Wyobraźmy sobie układy w postaci wielkich beczek wypełnionych po brzegi kłaczkowatą, płynną masą, która pozwala przyjrzeć się przyspieszonej ewolucji genów kodujących enzymy, spowodowanej dopływem związków chemicznych, z którymi bakterie muszą sobie radzić. Trudno? Wcale nie. A jednocześnie uzyskujemy cenne źródło informacji dotyczącej pozyskiwania szczepów bakteryjnych najefektywniejszych w procesach oczyszczania. Azot i jego związki są jednym z najniebezpieczniejszych wrogów czystych wód. Ich nadmiar, często w połączeniu z nadwyżką związków fosforu, powoduje tak częste w ciepłych miesiącach zakwity wód. Żadna przyjemność taplać się w „kwitnącej”, zielonej sadzawce na Mazurach. Stąd, choćby dla samej przyjemności kąpieli w czystej wodzie, usunięcie nadmiaru azotu to ważny krok.
Żelowe puzzle
Pierwszym etapem w mojej pracy jest izolacja całkowitego DNA komórek bakteryjnych, które stanowi matrycę do późniejszej amplifikacji (powielania). Mini-probówka, zestaw odczynników, trochę cierpliwości i... już. Gdybyście widzieli, jak mi się na początku ręce trzęsły!
Zmagania z materiałem, który przywiozłam z Polski, zaczęły się od najbardziej „pierwotnego” z narzędzi biologii molekularnej – reakcji łańcuchowej polimerazy, czyli PCR. Metoda, mająca na celu zwielokrotnienie ilości fragmentów interesującego nas genu o określonej długości, oparta jest na najbardziej podstawowej reakcji enzymatycznej żywej komórki – na replikacji. Używając fragmentów kwasu nukleinowego o znanej sekwencji, będących starterami reakcji, o strukturze homologicznej, czyli przystającej do badanej nici, można powielić miliardy razy fragment DNA znajdujący się między nimi (rys. 1). Reakcja PCR umożliwia przeprowadzenie eksperymentu na genach pochodzących z materiału, którego w pierwotnej fazie badań mieliśmy minimalne ilości. Uzyskane kopie genu stanowią substrat dalszych doświadczeń.
Jest druga w nocy. Oczy mi się kleją, ale dzielnie czekam na wynik reakcji PCR. No, wreszcie koniec! Dzięki uzyskaniu wielokrotnych kopii genu, możliwa jest jego separacja techniką elektoroforezy w żelu agarozowym. Trochę nieuwagi albo zmęczenia i... Plask! Żel spada na ziemię. Miałam rękawiczki mokre od buforu, więc teraz mam żelowe puzzle. Ech, masz ci los! Robimy nowy żel. Przygotowując żel dodaje się bromku etydyny – substancji umożliwiającej wizualizację molekuł DNA w świetle ultrafioletowym, po uprzednim wbudowaniu się bromku w podwójną helisę. Bromek jest jednak rakotwórczy, bez lateksowych rękawiczek ani rusz. Żel, zanurzony w buforze (tym samym, przez który rękawiczki się ślizgają) przewodzącym prąd, umieszcza się między elektrodami. DNA, jako cząstka kwasu, jest naładowana ujemnie, dlatego w polu elektrycznym wędruje w kierunku dodatniego bieguna. Hurra! Mam różowe prążki w żelu! Czas wreszcie iść spać. Właściwa „zabawa” dopiero się zacznie.
„Odciski palców”
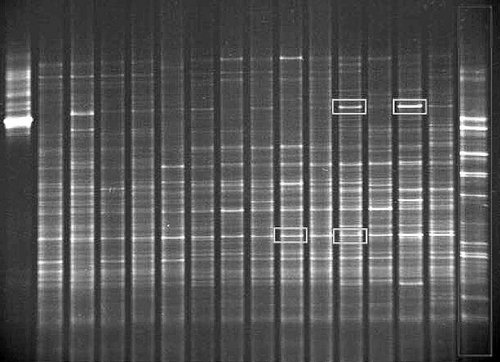
W chwili uzyskania wielokrotnych kopii genów bakteryjnych, używanych powszechnie do badań filogenetycznych – genów rybosomalnego DNA oraz genów enzymów związanych z utlenianiem amoniaku, jako związku szkodliwego w środowisku – zaczął się najciekawszy etap moich badań. Estończycy są „szczęśliwymi posiadaczami” sprzętu do specyficznego rodzaju elektroforezy – elektroforezy w gradiencie denaturacji (DGGE). W tej technice matriks dla produktów wcześniejszej reakcji PCR jest żel poliakrylamidowy, w którym pionowo lub poziomo rozłożona jest zmienna zawartość związków powodujących rozłączenie podwójnej helisy w cząstkach DNA. Pierwotnie system ten zaprojektowano do wykrywania mutacji, dzięki czemu w genetyce mikroorganizmów można go wykorzystać do porównywania sekwencji kwasu nukleinowego w tych samych genach różnych bakterii. Różnice w sekwencji nukleotydów warunkują odmienną ruchliwość cząstek w żelu denaturującym, zatrzymując je na innych wysokościach, odpowiadających różnemu stężeniu denaturanta. Dzięki temu uzyskujemy charakterystyczny dla próbki obraz „świecących prążków”. Porównując takie „odciski palców” poszczególnych próbek można z powodzeniem obserwować zachodzące w czasie zmiany w składzie grup osadu czynnego, nie tylko w jednym reaktorze, ale również w różnych typach bioreaktorów, co pozwala znaleźć bakterie odpowiedzialne za najefektywniejszy rozkład amoniaku.
Po każdym kroku procedury – wylanie żelu, przygotowanie buforu, wstawienie próbek – muszę zrobić obowiązkową przerwę. Elektroforeza będzie rozwijać DNA przez ponad dwanaście godzin, dlatego staram się wstawiać ją w nocy. Nie muszę wtedy zrywać się skoro świt. Chyba żaden z moich kolegów po fachu tego nie lubi. Po drugiej kawie wiszę nad pojemnikiem, gdzie żel moczy się w bromku etydyny. Zaraz sprawdzę, czy moje siedzenie do nocy w laboratorium się opłaciło. Zdecydowanie tak! (rys. 2)
Pojedyncze, ładnie oddzielone od reszty „odcisku palca” prążki, to jeden „typ DNA”, o takich samych wymogach co do warunków denaturacji. Najprawdopodobniej świadczy to o obecności jednego rodzaju bakterii. Pytanie: co to za bakterie? Jak można się dowiedzieć, które mikroorganizmy utylizują amoniak szybciej niż inne dominujące w próbce? Tu wkracza na scenę osławione w ostatnich latach sekwencjonowanie genów. Świecący na różowo intensywniej od innych prążek wycina się z żelu i wypłukuje z niego DNA, a następnie oczyszcza i przygotowuje kolejną reakcję PCR, tym razem z innym typem nukleotydów, które pozwolą odczytać sekwencję DNA, używając do tego celu sekwenatora – podłączonej do prądu „szafki” z kolejnym żelem poliakrylamidowym. Uzyskaną w wyniku sekwencjonowania kolejność ułożenia zasad azotowych wprowadza się do bazy danych (cóż uczony zrobiłby bez bezbrzeżnego morza informacji, jakim jest Internet!). Jeżeli mikroorganizm był wcześniej odnaleziony, uzyskamy informację o jego nazwie lub miejscu znalezienia. Jeśli nie, jesteśmy szczęśliwymi odkrywcami sekwencji genu kolejnego mikroskopijnego mieszkańca Ziemi.
Szukamy podstaw
Praca jest dość mozolna. Aby uzyskać wiarygodne wyniki, które potem będę mogła wykorzystać lub publikować, potrzebuję wielokrotnych powtórzeń moich analiz. Niejeden żel spadnie mi na podłogę i niejedna elektroforeza „rozmaże się” w świetle ultrafioletowym, zanim będę zadowolona z rezultatów badań. Ale te noce spędzone w laboratorium i godziny surfowania po Internecie w poszukiwaniu pojedynczej informacji, bez której nie ruszę z miejsca, mają swój urok. Robię to, co lubię i poznaję świat na poziomie „mikro” sprawdzając własnymi rękami reguły, które nim rządzą. Patrzę, słucham i uczę się. Uczę się pasji i radości z tego, co robię na co dzień. Chociażby po to warto współpracować z innymi, nie tylko za granicą. Pracuję teraz nad materiałem, który może być ze wszech miar użyteczny nie tylko naukowo, ale technologicznie i „życiowo”. Nie należąc do Greenpeace też można działać na rzecz hasła: „Save the planet”.
Współpraca z Estończykami najprawdopodobniej zaowocuje publikacją, ale wiedza, którą tam zdobyłam, jest dużo ważniejsza. Porównywanie, szukanie, analiza mnóstwa danych, noce spędzane w laboratorium i godziny ślęczenia przed komputerem to aktualnie życie badaczy.
Kiedyś wydawało mi się, że to jest coś w rodzaju „sztuki dla sztuki”. Zagłębiając się w ten dziwny naukowy świat, odkrywa się, że bez znajomości tego, co udaje nam się odkryć w laboratorium, są nikłe szanse na rozwój wszelkiego rodzaju technologii i przemysłu. Życie w cywilizowanym świecie bywa łatwiejsze dzięki znajomości podstaw. My szukamy tych podstaw. Bo przecież nieważne, jak bardzo skomplikowane jest zwinięcie helisy DNA, gdy zacznie się od podstaw, wszystko stanie się proste.


Komentarze
Tylko artykuły z ostatnich 12 miesięcy mogą być komentowane.